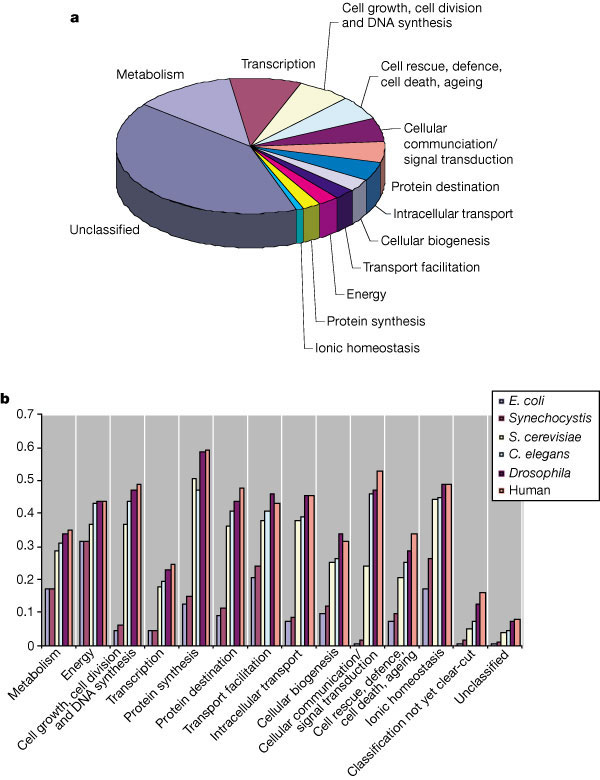
On utilise les logiciels de comparaison de séquence pour rechercher les orthologues dans chaque espèce.
Comment différencier orthologue de paralogue? On s'attend à ce que les orthologues soient les plus semblables (meilleur score BLAST).
Il existe des protocoles selectionnant des groupes d'au moins 3 protéines ayant réciproquement les meilleurs score (cf COG).
Ces procédures permettent de s'abstraire du niveau de ressemblance entre orthologues.
Permettent d'identifier les grandes fonctions ancestrales, communes à certaines classes d'organismes.
Un groupe d'ortholoue très répandu (p. ex. eucaryotes, archae et eubactéries) est un bon candidat pour des travaux expérimentaux.
| Traduction | Métabolisme énergétique | Transcription |
| Processus cellulaires | Pur., pyr., nucléosides et nucléotides | Métabolisme acides gras et phospholipides |
| Biosynthèses aminoacides | Métabolisme intermédiaire central | Protéines de transport et de liaison |
| Enveloppe cellulaire | Réplication | Fonctions Régulatoires |
| Biosynthèse cofacteurs, gpe prosthétiques et transporteurs | Protéines ribosomiques | Autres |
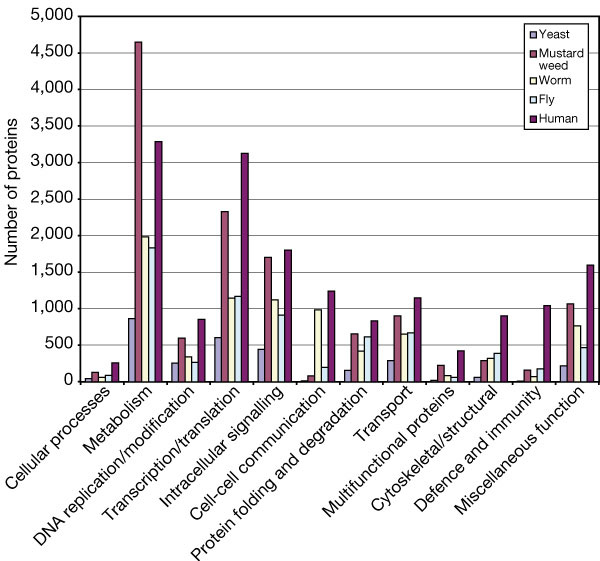
Functional categories in eukaryotic proteomes. The classification categories were derived from functional classification systems, including the top-level biological function category of the Gene Ontology project (GO; see http://www.geneontology.org).
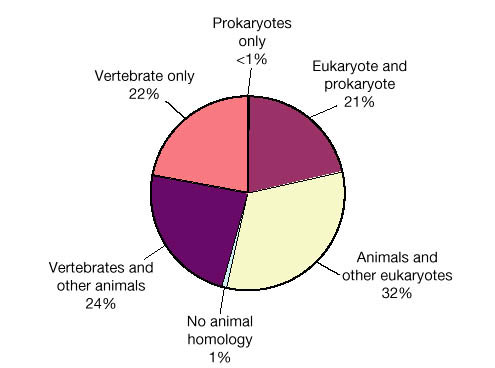
Distribution of the homologues of the predicted human proteins. For each protein, a homologue to a phylogenetic lineage was considered present if a search of the NCBI nonredundant protein sequence database, using the gapped BLASTP program, gave a random expectation (E ) value of 0.001. Additional searches for probable homologues with lower sequence conservation were performed using the PSI-BLAST program, run for three iterations using the same cut-off for inclusion of sequences into the profile328.
Protéome d'Arabidopsis