Promoteurs et région 5'
Une séquence promotrice flanque l'exon 5'.
Il existe trois types de promoteurs eucaryotes, pour les trois
types d'ARN polymérase les reconnaissant:
Pol-I, Pol-II et Pol-III. Les promoteurs Pol-I se
trouvent face aux ARNr 18S et 28S. Les promoteurs Pol-II se
trouvent face aux ARNm. Les promoteurs Pol-III se
trouvent face aux ARNt et ARNr 5S.
Les promoteurs de type Pol-II sont généralement les plus
intéressants car ils signalent les gènes protéiques. Ces promoteurs
contiennent une boîte TATA et au moins une autre séquence importante
en amont.
On peut la schématiser
ainsi (TATA=boîte TATA, INR= Initiateur):
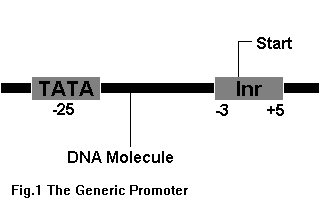
Facteurs protéiques liant le promoteur, juste avant
l'initiation de la transcription. Cas typique, mais nullement
général, les facteurs de variant considérablement suivant les gènes
et l'environnment. On connait plus de 2000 facteurs de transcription.

La boîte TATA
La majorité des promoteur de gènes protéiques eucaryotes
contiennent une boîte TATA.
-
La boîte TATA est trouvée à 25-30 paires de bases en amont du site
de départ de transcription (TSS). Une position relativement constante dans les promoteurs eucaryotes.
C'est le seul
signal dans le promoteur se trouvant à une distance définie du TSS.
- La sequence d'environ 8 paires de bases contient pratiquement que des adenines et thymines,
et tend a être encadrée par des séquences riches en guanine
et cytosine, ces dernières pouvant participer à la fonction du promoteur.
- La boîte TATA est très semblable à la séquence TATA des
procaryotes, la position mise à part (-10 chez les procaryotes).
- TATA consensus: GTATAAAAGGCGGGG (mais beaucoup
de variation)
Le consensus de la TATA box est faible et cet élément est même
absent dans de nombreux promoteurs.
General eukaryotic TATA-box model derived from 860 unrelated promoter sequences:
| Position |
1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
10 |
11 |
12 |
| % A |
19.4 |
23.4 |
5.0 |
83.5 |
4.4 |
89.2 |
71.0 |
84.8 |
45.0 |
35.7 |
15.5 |
18.5 |
| % C |
22.7 |
34.0 |
11.0 |
1.3 |
3.3 |
0.8 |
0.8 |
2.9 |
3.4 |
14.0 |
36.5 |
37.0 |
| % G |
26.5 |
30.8 |
4.5 |
1.4 |
0.9 |
1.7 |
0.5 |
9.5 |
16.4 |
38.4 |
36.3 |
30.4 |
| % T |
31.4 |
11.7 |
79.5 |
13.9 |
91.4 |
8.4 |
27.7 |
2.8 |
35.2 |
11.8 |
11.7 |
14.1 |
| Consensus |
|
|
T |
A |
T |
A |
W |
A |
D |
R |
|
|
L'Initiateur
L'initiateur (INR), se trouve près du site de début de transcription,
entre les positions -3 et +5. Il y a peu ou pas de similarité entre les
initiateurs de différents promoteurs, toutefois, la première base du
mRNA transcrit tend à être un A, souvent flanqué de pyrimidines.
La RNA Pol II peut parfois initier la transcription avec l'INR
seul, dans des promoteurs simples sans boîte TATA.
Les éléments en amont du promoteur
Séquences courtes de 6-20 nt affectant généralement l'
efficacité de l'initiation de la transcription.
- La boîte CCAAT
- Sp1 box
- CRE
- AP2 box
etc..
Document: Les TATA Box Binding Proteins (TBP)
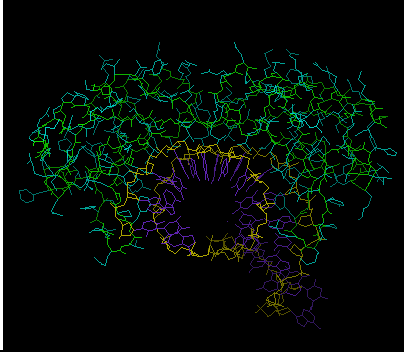 TATA Binding Proteins
The TBP is a small protein of about 3000 Daltons in mass. It is the first factor to make contact with the DNA and is
known as the commitment factor, as once the TBP has bound to the TATA-Box, the promoter is transcribed.
TATA Binding Proteins
The TBP is a small protein of about 3000 Daltons in mass. It is the first factor to make contact with the DNA and is
known as the commitment factor, as once the TBP has bound to the TATA-Box, the promoter is transcribed.
As the TATA-Box is a fixed distance away from the start point, it's recognition by the T F II D is vital in positioning
the BIC.
The TBP binds into the minor groove of the double stranded DNA molecule and consists of 2 similar domains (40%
homologous) which are highly conserved in all eukaryotes, and a very variable N-Terminal region. When bound to
the DNA, the 2 similar domains 'straddle' one side of the DNA molecule, which binds to their inner surface. The
N-Terminal end binds to the other proteins of the BIC.
The
picture above, shows the TBP, bound to the DNA. The 2 similar domains (pale blue and green) straddle the DNA
(dark blue).
There is more than a passing similarity between the TBP and the sigma factor of bacterial promoters, and the TBP
has a 30% homology to it. The TBP is highly conserved in all eukaryotes.
The TBP is the only BTF to bind specifically to the DNA.
The TBP covers 1 turn of the DNA molecule between positions -37 and -25.
By Matthew I. Walton, E-mail M.I.Walton@stud.man.ac.uk.
University of Manchester (1997).
Les ilots CpG
Les ilôts CpG sont des zones riches en dinucléotide CG, fréquemment associées aux régions 5' des gènes de vertébrés
- L'ilôt s'étend sur le promoteur et l'exon 1 (ou 1 et 2)
- Fréquence attendue du dinucléotide CpG = 4% (0.21x0.21), mais fréquence observée:
un cinquième de cette valeur. Pourquoi?
- Méthylation naturelle des CpG et réparation en TpG
- Au niveau des premiers exons: protection des CpG. Donc Fréquence normale.
- Typiquement 1-2kb de longueur. Environ about 70% G et C (contre
40% dans le reste du génome humain)
-
Les îlots CpG sont associés à tous les gènes
housekeeping (constitutifs) et à 40% des gènes tissu-spécifiques
Extrait de l'article de Nature sur le génome humain
|
The dinucleotide CpG is notable because it is greatly
under-represented in human DNA, occurring at only about one-
fth of the roughly 4% frequency that would be expected by simply
multiplying the typical fraction of Cs and Gs (0.21 ´ 0.21). The
deficit occurs because most CpG dinucleotides are methylated on
the cytosine base, and spontaneous deamination of methyl-C
residues gives rise to T residues. (Spontaneous deamination of
ordinary cytosine residues gives rise to uracil residues that are
readily recognized and repaired by the cell.) As a result, methyl-
CpG dinucleotides steadily mutate to TpG dinucleotides. However,
the genome contains many `CpG islands' in which CpG dinucleo-
tides are not methylated and occur at a frequency closer to that
predicted by the local GC content. CpG islands are of particular
interest because many are associated with the 5' ends of genes
|


 TATA Binding Proteins
The TBP is a small protein of about 3000 Daltons in mass. It is the first factor to make contact with the DNA and is
known as the commitment factor, as once the TBP has bound to the TATA-Box, the promoter is transcribed.
TATA Binding Proteins
The TBP is a small protein of about 3000 Daltons in mass. It is the first factor to make contact with the DNA and is
known as the commitment factor, as once the TBP has bound to the TATA-Box, the promoter is transcribed.